[Data Analysis] 차원축소 기반 이상 탐지
PCA, t-SNE을 이용한 이상탐지 구현해보기
[Data Analysis] 차원축소 기반 이상 탐지
PCA 이상 탐지
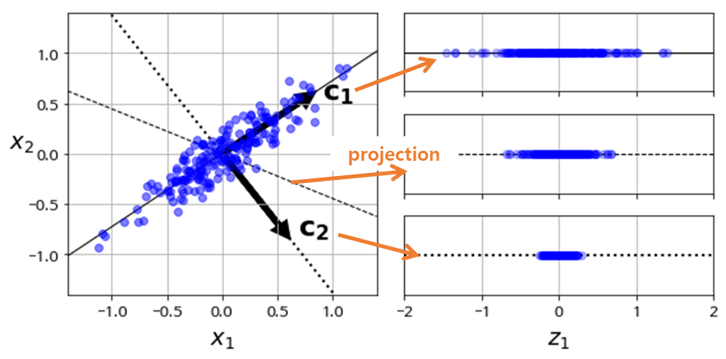
PCA(Principal Component Analysis)란 변수에서 가장 가까운 초평면을 구한 다음, 초평면을 투영(Projection)하여 차원을 축소하는 기법입니다.
AutoEncoder의 작동원리인 정보를 압축하고 복원하면서 데이터를 실제와 가깝게 복원시키기 위해 정상 데이터의 패턴을 학습하고 비 정상인 데이터는 복원 패턴을 배우지않으며 비정상인 데이터를 찾는 것과 비슷합니다.
PCA는 다음과 같은 단계로 이루어집니다.
- 학습 데이터셋에서 분산이 최대인 축(axis)을 찾습니다.
- 이렇게 찾은 첫번째 축과 직교(orthogonal)하면서 분산이 최대인 두 번째 축을 찾습니다.
- 첫 번째 축과 두 번째 축에 직교하고 분산을 최대한 보존하는 세 번째 축을 찾습니다.
- 1~3과 같은 방법으로 데이터셋의 차원(특성 수)만큼의 축을 찾습니다.
- 장점
- 선택한 변수들의 해석이 용이함
- 고차원에서 저차원으로 줄일 수 있음
- 단점
- 변수간 상관관계를 고려하기 어려움
- 추출된 변수 해석 어려움
주의 : PCA는 정규화를 필수로 요구합니다.(아래 데이터는 정규화가 진행되어있습니다.)
1
2
3
4
5
from sklearn.decomposition import PCA
import pandas as pd
data = pd.read_csv('creditcard.csv')
data
| Time | V1 | V2 | V3 | V4 | V5 | V6 | V7 | V8 | V9 | ... | V21 | V22 | V23 | V24 | V25 | V26 | V27 | V28 | Amount | Class | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0.0 | -1.359807 | -0.072781 | 2.536347 | 1.378155 | -0.338321 | 0.462388 | 0.239599 | 0.098698 | 0.363787 | ... | -0.018307 | 0.277838 | -0.110474 | 0.066928 | 0.128539 | -0.189115 | 0.133558 | -0.021053 | 149.62 | 0 |
| 1 | 0.0 | 1.191857 | 0.266151 | 0.166480 | 0.448154 | 0.060018 | -0.082361 | -0.078803 | 0.085102 | -0.255425 | ... | -0.225775 | -0.638672 | 0.101288 | -0.339846 | 0.167170 | 0.125895 | -0.008983 | 0.014724 | 2.69 | 0 |
| 2 | 1.0 | -1.358354 | -1.340163 | 1.773209 | 0.379780 | -0.503198 | 1.800499 | 0.791461 | 0.247676 | -1.514654 | ... | 0.247998 | 0.771679 | 0.909412 | -0.689281 | -0.327642 | -0.139097 | -0.055353 | -0.059752 | 378.66 | 0 |
| 3 | 1.0 | -0.966272 | -0.185226 | 1.792993 | -0.863291 | -0.010309 | 1.247203 | 0.237609 | 0.377436 | -1.387024 | ... | -0.108300 | 0.005274 | -0.190321 | -1.175575 | 0.647376 | -0.221929 | 0.062723 | 0.061458 | 123.50 | 0 |
| 4 | 2.0 | -1.158233 | 0.877737 | 1.548718 | 0.403034 | -0.407193 | 0.095921 | 0.592941 | -0.270533 | 0.817739 | ... | -0.009431 | 0.798278 | -0.137458 | 0.141267 | -0.206010 | 0.502292 | 0.219422 | 0.215153 | 69.99 | 0 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 284802 | 172786.0 | -11.881118 | 10.071785 | -9.834783 | -2.066656 | -5.364473 | -2.606837 | -4.918215 | 7.305334 | 1.914428 | ... | 0.213454 | 0.111864 | 1.014480 | -0.509348 | 1.436807 | 0.250034 | 0.943651 | 0.823731 | 0.77 | 0 |
| 284803 | 172787.0 | -0.732789 | -0.055080 | 2.035030 | -0.738589 | 0.868229 | 1.058415 | 0.024330 | 0.294869 | 0.584800 | ... | 0.214205 | 0.924384 | 0.012463 | -1.016226 | -0.606624 | -0.395255 | 0.068472 | -0.053527 | 24.79 | 0 |
| 284804 | 172788.0 | 1.919565 | -0.301254 | -3.249640 | -0.557828 | 2.630515 | 3.031260 | -0.296827 | 0.708417 | 0.432454 | ... | 0.232045 | 0.578229 | -0.037501 | 0.640134 | 0.265745 | -0.087371 | 0.004455 | -0.026561 | 67.88 | 0 |
| 284805 | 172788.0 | -0.240440 | 0.530483 | 0.702510 | 0.689799 | -0.377961 | 0.623708 | -0.686180 | 0.679145 | 0.392087 | ... | 0.265245 | 0.800049 | -0.163298 | 0.123205 | -0.569159 | 0.546668 | 0.108821 | 0.104533 | 10.00 | 0 |
| 284806 | 172792.0 | -0.533413 | -0.189733 | 0.703337 | -0.506271 | -0.012546 | -0.649617 | 1.577006 | -0.414650 | 0.486180 | ... | 0.261057 | 0.643078 | 0.376777 | 0.008797 | -0.473649 | -0.818267 | -0.002415 | 0.013649 | 217.00 | 0 |
284807 rows × 31 columns
1
2
# Time, Amomunt 변수 제거
data.drop(['Time', 'Amount'], axis=1, inplace=True)
1
2
pca = PCA()
pca.fit(data.drop(['Class'], axis=1))
PCA()In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
PCA()
1
2
3
4
5
6
7
8
9
10
11
# 차원축소 주성분 갯수 확인
features = range(pca.n_components_)
feature_df=pd.DataFrame(data=features,columns=['pc_feature'])
# 설명력 확인
variance_df=pd.DataFrame(data=pca.explained_variance_ratio_,columns=['variance'])
variance_df["cumulative_variance"] = variance_df["variance"].cumsum()
# 전체 분산에 95% 이상 설명 가능(feature 22개 채택)
pc_feature_df=pd.concat([feature_df,variance_df],axis=1)
pc_feature_df
| pc_feature | variance | cumulative_variance | |
|---|---|---|---|
| 0 | 0 | 0.124838 | 0.124838 |
| 1 | 1 | 0.088729 | 0.213567 |
| 2 | 2 | 0.074809 | 0.288376 |
| 3 | 3 | 0.065231 | 0.353608 |
| 4 | 4 | 0.061990 | 0.415598 |
| 5 | 5 | 0.057756 | 0.473354 |
| 6 | 6 | 0.049799 | 0.523153 |
| 7 | 7 | 0.046417 | 0.569570 |
| 8 | 8 | 0.039275 | 0.608845 |
| 9 | 9 | 0.038579 | 0.647423 |
| 10 | 10 | 0.033901 | 0.681325 |
| 11 | 11 | 0.032488 | 0.713812 |
| 12 | 12 | 0.032233 | 0.746045 |
| 13 | 13 | 0.029901 | 0.775946 |
| 14 | 14 | 0.027262 | 0.803208 |
| 15 | 15 | 0.024984 | 0.828192 |
| 16 | 16 | 0.023473 | 0.851665 |
| 17 | 17 | 0.022860 | 0.874526 |
| 18 | 18 | 0.021563 | 0.896088 |
| 19 | 19 | 0.019339 | 0.915427 |
| 20 | 20 | 0.017556 | 0.932983 |
| 21 | 21 | 0.017137 | 0.950120 |
| 22 | 22 | 0.012689 | 0.962809 |
| 23 | 23 | 0.011936 | 0.974745 |
| 24 | 24 | 0.008842 | 0.983587 |
| 25 | 25 | 0.007567 | 0.991153 |
| 26 | 26 | 0.005301 | 0.996455 |
| 27 | 27 | 0.003545 | 1.000000 |
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
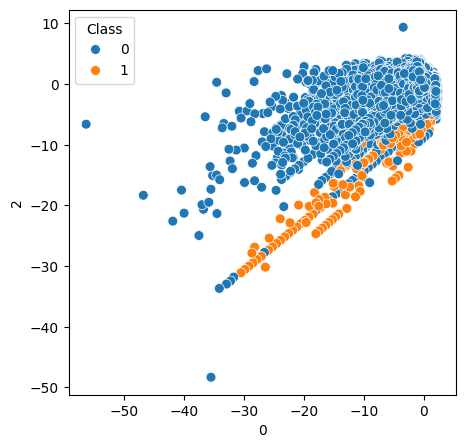
x = data.drop(['Class'], axis=1)
y = data['Class']
pca = PCA(n_components=22)
x_pca = pca.fit_transform(x)
pc_df=pd.DataFrame(x_pca,columns=[f'{i}' for i in range(22)]).reset_index(drop=True)
pc_df=pd.concat([pc_df,y],axis=1)
plt.rcParams['figure.figsize'] = [5, 5]
sns.scatterplot(data=pc_df,x='0',y='2',hue=y, legend='brief', s=50, linewidth=0.5);
t-SNE 이상 탐지
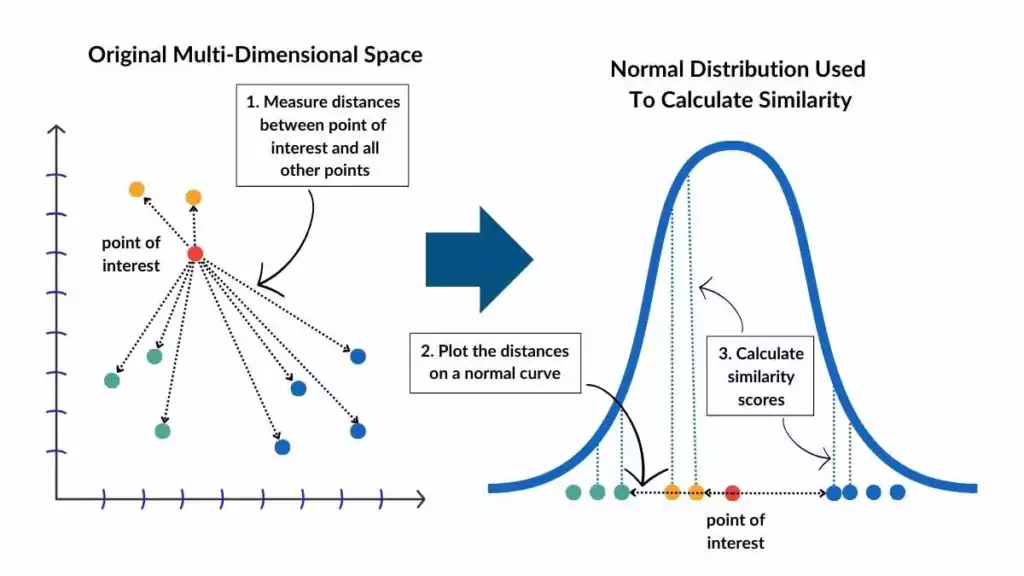
t-SNE(t-distributed Stochastic Neighbor Embedding)는 비선형 차원 축소 기법으로 t-분포를 사용하여 데이터 유사도를 계산하여 유사도가 낮은 데이터는 멀리 떨어뜨려 거리를 최대한 보존합니다.
🔹 t-SNE 동작 과정
고차원 공간에서 데이터 포인트 간 확률적 유사도 계산
저차원 공간에서 랜덤하게 초기화된 점들의 분포 생성
저차원 공간에서도 유사한 확률 분포를 유지하도록 위치 조정
최적화 수렴 후 최종 시각화 결과 생성
- 장점
- PCA 대비 클러스터링을 발견하기 쉬움
- 비선형 관계의 데이터를 군집화 가능
- 단점
- 대량의 데이터에서 속도가 느림 (O(n²) 복잡도)
- 매번 돌릴 때마다 다른 시각화 결과가 도출
1
2
3
4
5
6
7
from sklearn.manifold import TSNE
from sklearn.model_selection import train_test_split
# 데이터 갯수 줄이기(약 30만개의 데이터를 넣으면 시간이 오래걸립니다.)
df_reduced, _ = train_test_split(data, train_size=0.03, stratify=data['Class'], random_state=42)
df_reduced['Class'].value_counts()
| count | |
|---|---|
| Class | |
| 0 | 8529 |
| 1 | 15 |
1
2
3
4
5
# t-SNE 2차원 축소(t-SNE는 4차원 이상으로 변환할 수 없습니다.)
tsne_np = TSNE(n_components = 3, random_state=42).fit_transform(df_reduced.drop(['Class'], axis=1))
tsne_df = pd.DataFrame(tsne_np, columns = [f'{i}' for i in range(3)])
tsne_df
| 0 | 1 | 2 | |
|---|---|---|---|
| 0 | -2.370444 | -12.131145 | 19.459503 |
| 1 | -18.261276 | 0.444234 | -0.662202 |
| 2 | 5.660702 | -2.703253 | -14.699653 |
| 3 | 0.748979 | 4.191403 | 4.901437 |
| 4 | -6.995453 | -8.831751 | -17.670755 |
| ... | ... | ... | ... |
| 8539 | 11.484577 | 18.109447 | -3.144772 |
| 8540 | 11.609701 | 14.841992 | 8.453781 |
| 8541 | -6.828044 | -9.141667 | -16.934467 |
| 8542 | 2.451750 | -3.080299 | -7.047642 |
| 8543 | 7.451354 | -21.366283 | -0.024991 |
8544 rows × 3 columns
1
2
3
4
5
6
7
8
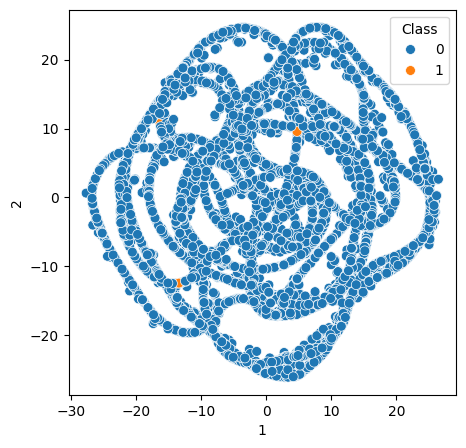
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
y = df_reduced['Class'].reset_index(drop=True)
plt.rcParams['figure.figsize'] = [5, 5]
sns.scatterplot(data=tsne_df,x='1',y='2',hue=y, legend='brief', s=50, linewidth=0.5);
참고자료
이 기사는 저작권자의 CC BY 4.0 라이센스를 따릅니다.